研究内容
私たちの研究室では、計算物理化学的アプローチと情報科学的アプローチを組み合わせることで生命現象を理解することを目標とし、研究を進めています。計算物理化学では分子動力学シミュレーションや量子化学計算を用い、情報科学では機械学習や構造インフォマティクスを活用することで生体分子の設計や制御方法を開発し、最終的には創薬へとつなげることを目指します。
研究テーマ
生体高分子の分子動力学シミュレーションによるダイナミクスの解明
- MDシミュレーションによるGPCRのダイナミクスとシグナル伝達機構の解明
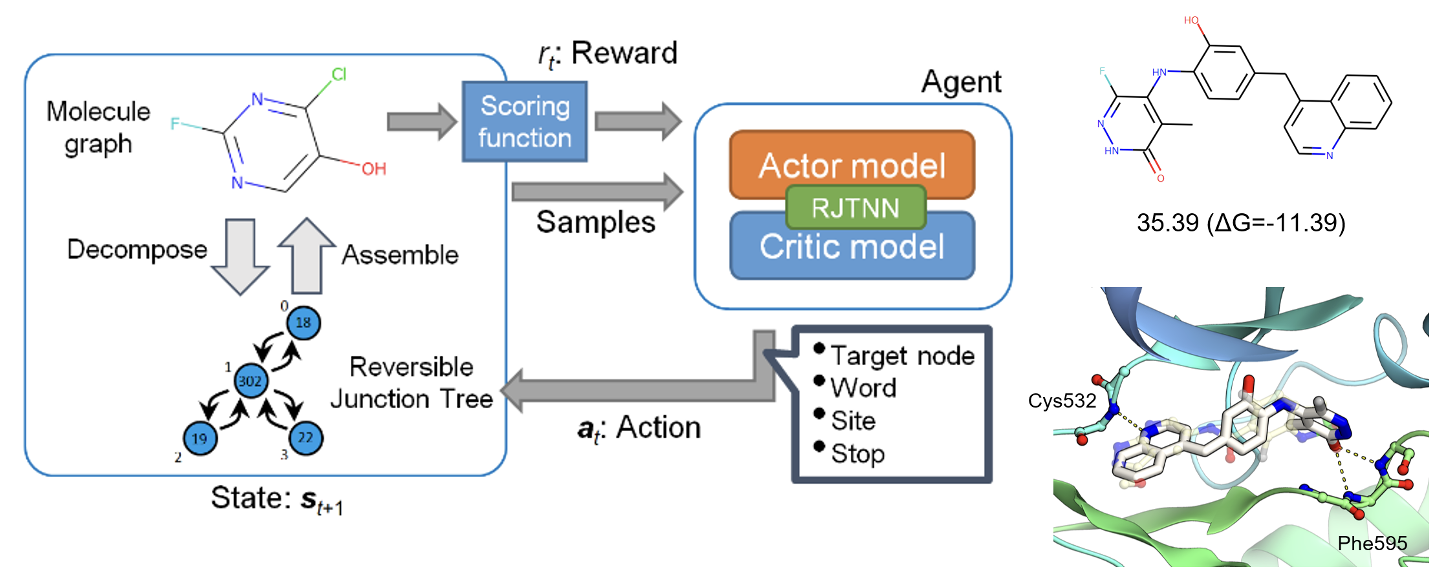
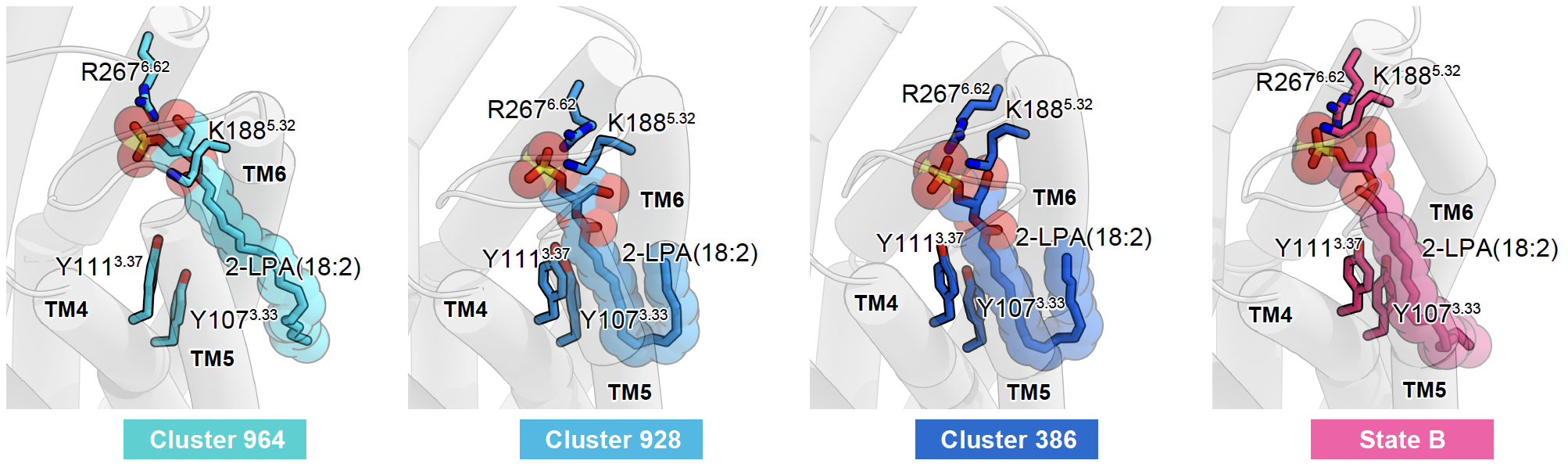
LPA6受容体の側方アクセス機構の検証
AlphaFoldを用いたタンパク質構造予測とその構造情報の応用
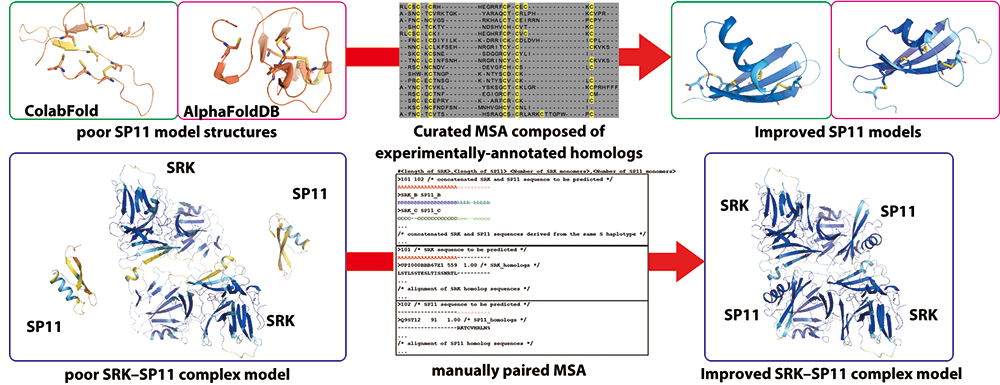
- 精選したMultiple sequence alignment (MSA)とColabFoldを用いることでアブラナ科の自家不和合性を制御するハプロタイプ特異的なSRK-SP11複合体を高信頼度で予測 Comprehensive computational analysis of the SRK–SP11 molecular interaction underlying self-incompatibility in Brassicaceae using improved structure prediction for cysteine-rich proteins
予測構造と量子化学シミュレーションを用いた、新規生合成酵素の反応機構解明
-
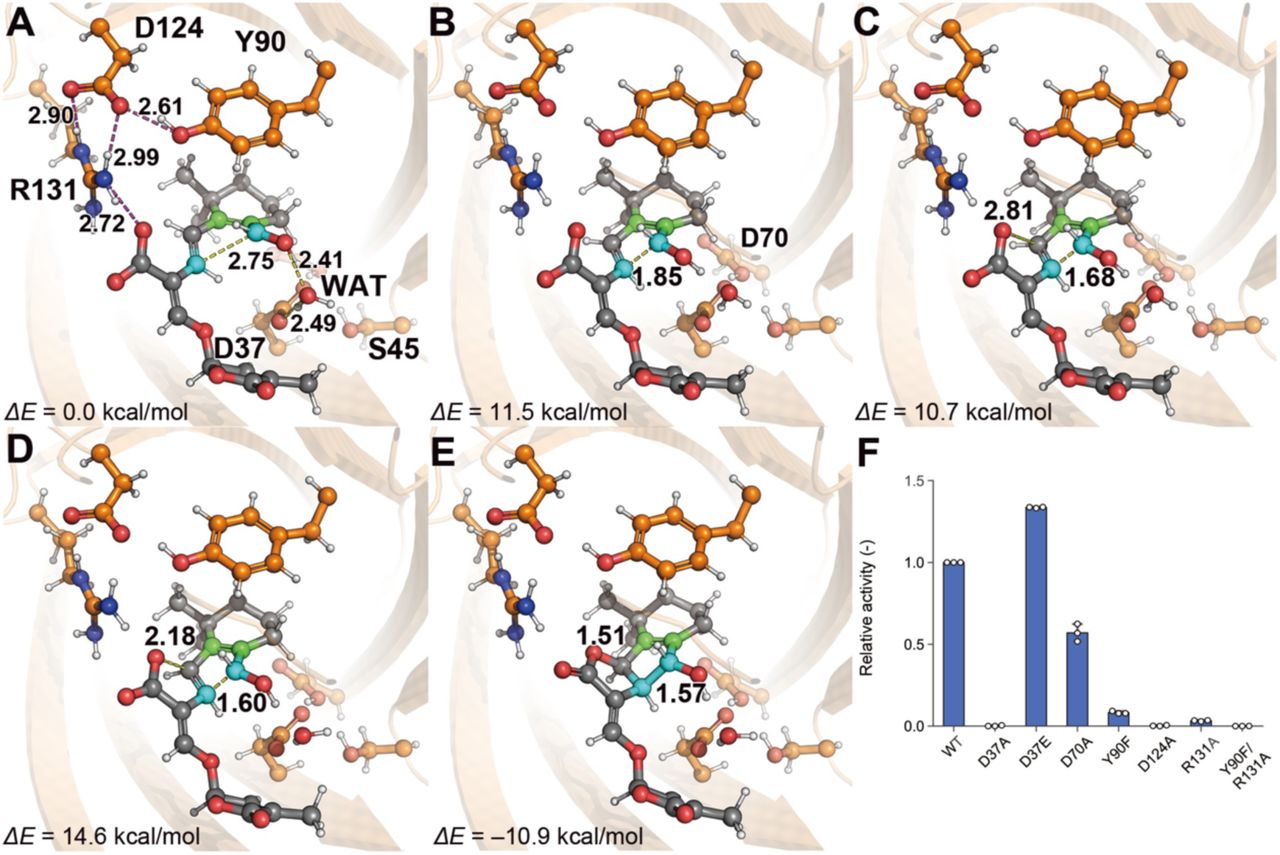
AlphaFold2の予測構造とMDシミュレーション、QM/MM(ONIOM)法を組み合わせたストリゴラクトンの新規生合成機構の解明 Insights into stereoselective ring formation in canonical strigolactone: Discovery of a dirigent domain-containing enzyme catalyzing orobanchol synthesis
-
PHBHの芳香族求電子置換反応のQM/MMシミュレーション 参考:ONIOM Tutorial -PHBHでの水酸化反応を使った練習-
分子シミュレーションと深層学習の融合による生体高分子のダイナミクスの解析手法の開発
- 深層学習の構造生物学への応用
- グラフニューラルネットワークを用いたタンパク質のプロパティ予測
- 深層学習の構造ベース低分子・中分子創薬への応用